안녕하세요. 이번에는 LEA-Net이라고 불리는 color reconstruction 기반 anomaly detection 논문에 대해서 리뷰해보겠습니다.
먼저 해당 논문을 읽게된 계기는 현재 작물질병검출 관련하여 논문을 작성중인데 작물질병검출 분야에서는 color reconstruction 기반의 이상검출을 많이 사용되므로 읽어보면 좋을거같아서 읽게되었습니다.
논문을 읽기전에 먼저 이상검출에 대해서 이야기를 해보겠습니다. 일반적으로 이상검출이라고하면 크게 지도학습기반 방법론들과 비지도학습 기반의 방법론들로 나뉘게 됩니다.
각기 방법론마다 장단점이 명확히 존재하며, 간략하게만 얘기해보겠습니다.
먼저, 지도학습 기반의 방법론의 경우에는 명확하게 답이존재하고, class imbalance문제가 없는 경우에는 좋은 결과를 가지고 올 수 있습니다. 그러나, labeling cost가 많이들고, 사람이 정확하게 labeling을 하기 힘든 경우 labeling error가 존재하게 됩니다. 이와 더불어 이상데이터와 정상데이터간의 class imbalance가 존재하는 경우에 지도학습 기반의 모델들은 dominant 한 class에 오버피팅되는 경향이 있습니다.
지도학습 기반의 방법론의 이러한 단점을 보완하고자, 비지도학습기반의 방법론들도 많이 사용되고 있습니다. 비지도학습 기반의 방법론은 크게 reconstruction based와 representation 방법론으로 나뉘며, 기본적으로 학습데이터에 대해서 라벨링작업이 필요없다는 큰 이점을 가집니다. 그리고, 이상치의 경계가 모호한경우에도 효과적으로 적용이 가능하기 때문에 이상검출에서는 비지도학습이 좀 더 일반적인 접근방법으로 사용되고있습니다.
물론 일반적이지 않은 상황에서는 지도학습이 비지도학습보다 이점을가지는 경우도 있습니다. 예를들어 cvpr 2021년에 나왔던 하수처리장에서 실제로 취득한 데이터셋의 경우에는 비정상상황, 정상상황에 대해서 라벨을 제공하고, 비정상상황인 비율이 전체데이터의 거의 절반을 차지할정도로 비정상데이터와 정상데이터간의 밸런스가 잘 맞습니다. 즉, 클래스 불균형 문제가 없어서 지도학습 기반의 모델을 적용하기에 적합합니다.
이런 특수한 경우를 제외한 일반적인 경우 비지도학습이 좀 더 유리한게 사실이며, 산업현장에서 발생할 수 있는 이상치를 포함한 MVTec-AD 논문을 기점으로 많은 방법론들이 제안되어 왔습니다.
MVTec-AD 데이터셋은 물체의 shape이 크게 변하지 않는 정형데이터만을 다루지만, 작물질병검출 분야에서는 같은 클래스에 속하는 작물이더라도, 잎사귀의 모양이 조금씩 상이하기 때문에 상당히 다른 특성을 가집니다.
이런 맥락에서 MVTec-AD데이터셋을 다룬 논문들과 다르게 작물에서의 이상검출은 어떤식으로 진행이 되는지에 대한 전반적인 이해를 하기에 좋은 논문이라고 생각하여 해당 논문을 리뷰하게 되었습니다.
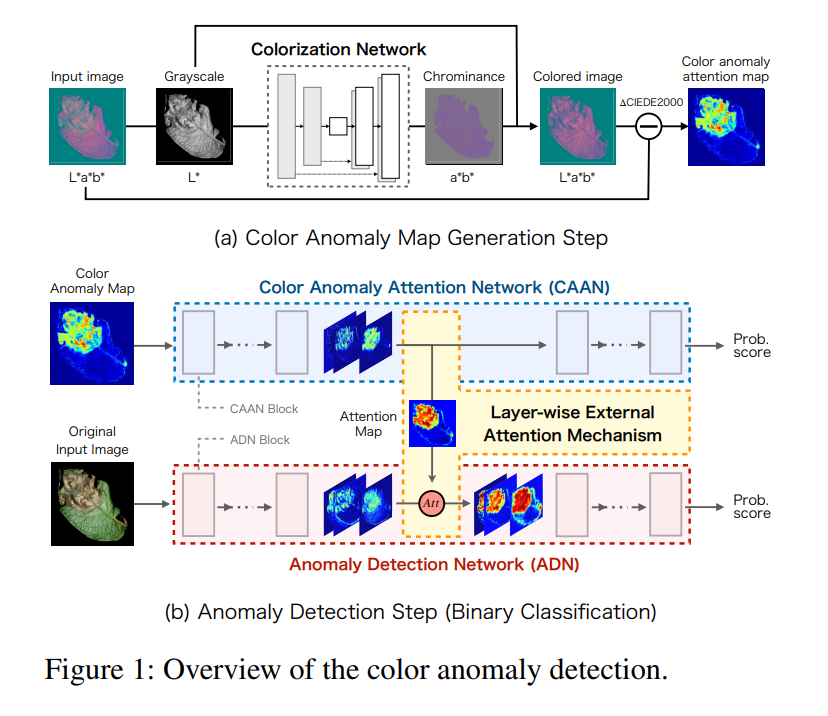
먼저, 해당 논문의 전반적인 파이프라인 입니다. 우선 (a)에 해당하는 color anomaly map generation step을 거쳐서 color reconstruction을 기반으로 attention map을 추출하여 (b)에다가 사용합니다.
좀 더 구체적으로, 인풋영상은 L*a*b로 들어가며, L에 해당하는 grayscale의 이미지가 인풋으로 들어갔을때, a, b을 predict하는 모델을 학습합니다. 그리고 이렇게 predicted된 a, b를 다시 L과 결합하여 L*a*b를 reconstuction하며, 이때, 이상치를 포함하는 픽셀의 경우에는 reconstruction이 잘 되지 않을 것 입니다. 이러한 정보를 이용하여 reconstruction된 영상과 원본영상의 ΔCIEDE2000 차이를 구하면 reconstruction이 잘 되지 않은 영역만이 남을 것이며, 해당 정보를 attention map으로 사용하게됩니다.
지금까지 설명한 color reconstruction네트워크를 학습하여 attention map을 구하는데까지는 비지도기반인데 논문을 계속 읽다보니 그림1 기준 아래쪽 네트워크인 CAAN과 ADN은 지도학습 기반인거 같네요..
단순히 ΔCIEDE2000만을 이용하여 일정한 쓰레드컷이상을 anomaly 픽셀로 분류하는게 사실 참 애매하기 때문에 해당 anomaly map을 attention map으로 활용하여 별도의 지도학습기반의 네트워크를 학습하는거 같습니다.
사실 학습은 이진분류이므로 그리 어렵지 않고요. 아래 그림처럼 모바일넷으로 피쳐를 추출하여 구성하였습니다.
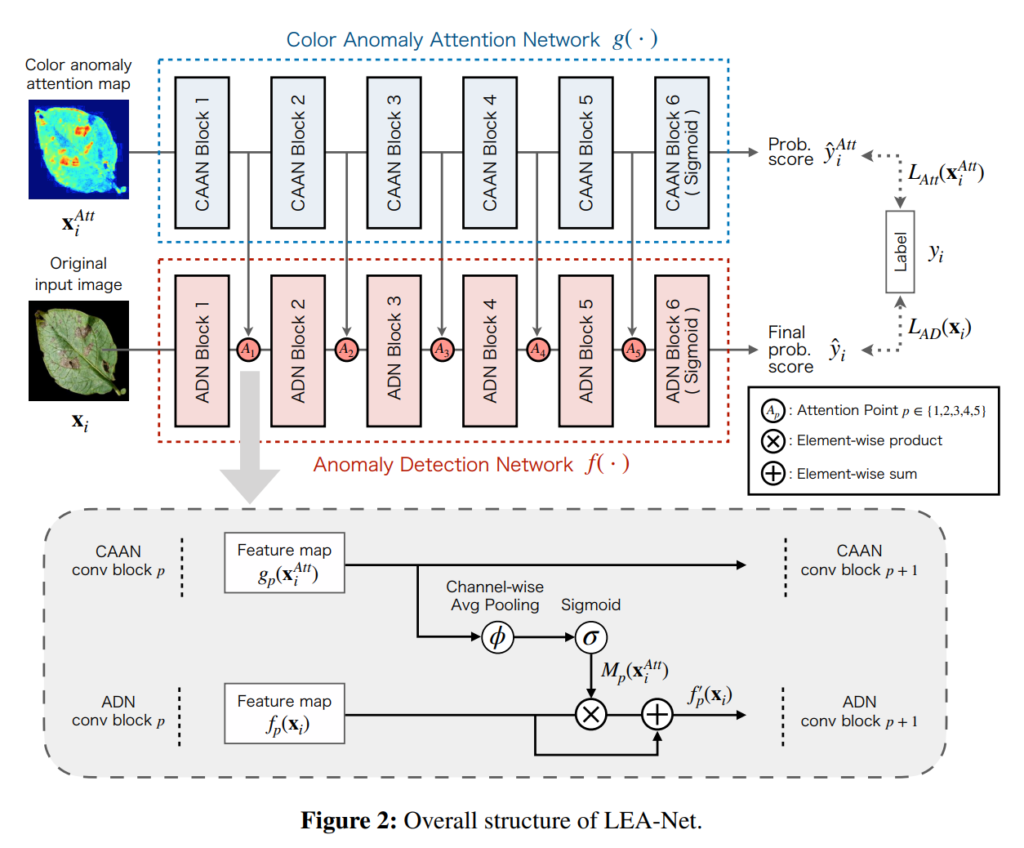
위의 그림을 이해해보자면, GT label이 존재하는 전형적인 이진분류 네트워크이며, 중간레벨 피쳐에서 sigmoid 함수를 통과시킨 피쳐값으로 아래쪽 피쳐들에 attention을 주는 것을 알 수 있습니다. Loss는 단순하게 L_AD와 L_att의 합으로 나타내어 지며, 학습하는 과정은 논문에서 formal하게 작성하기위해 이런저런 수식을 많이 사용하지만 결국엔 그냥 CNN 이진분류모델을 백프롭하여 학습시켰다는 이야기를 하고있네요.
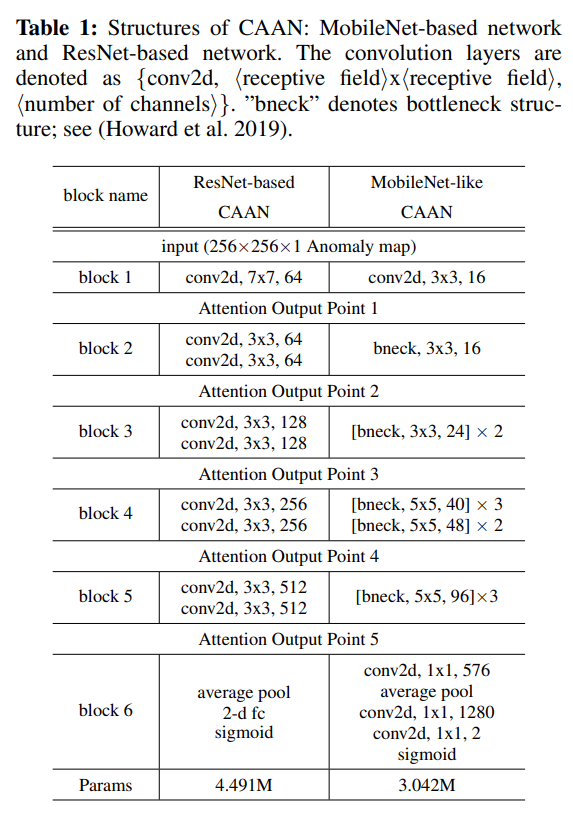
위의 테이블은 네트워크의 좀 더 상세한 정보입니다.

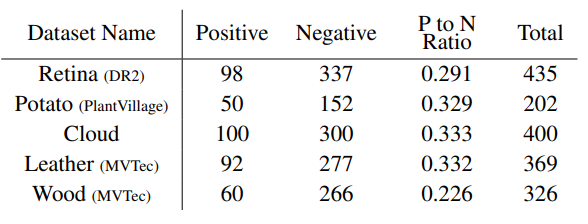
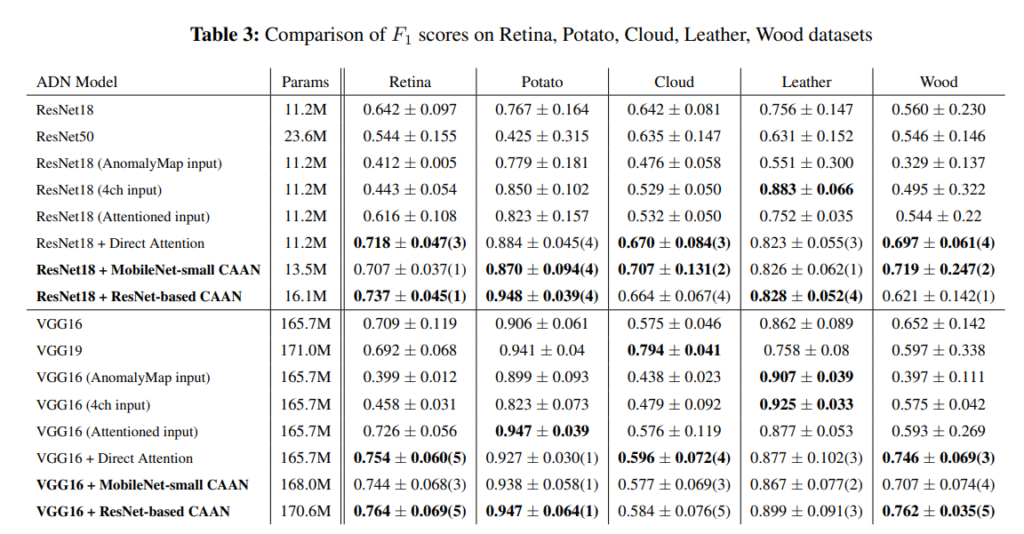
실험은 PlantVillage라는 식물데이터셋과 MVTec 등 총 4개의 데이터셋에서 일부 클래스를 랜덤샘플링하여 진행하였습니다. self-evaluation을 한것도 그렇고, 평가매트릭도 F1 score라서 다른방법론들과 좀 얼라인이 안맞는거 같습니다. 전체적인 성능위 위의 표와 같습니다.
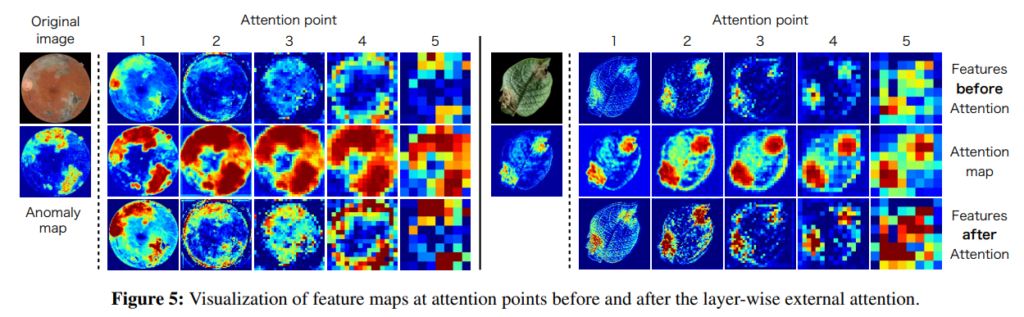
위의 그림은 attention이 적용된 이후 피쳐맵이 어떤식으로 보이는지 visualization한 모습입니다. (어떻게 visualization한지 나와있진 않네요…)
해당 논문으로 컨셉적으로 식물에 어떤식으로 anomaly detection 파이프라인을 적용할 것인지에 대한 감을 잡는데 도움이 된 논문이었으나, 역시 메이저학회 논문이 아니라 그런지 많이 부족한 논문이라고 생각이 드네요.
ΔCIEDE2000 차이를 계산하는게 Lab 스페이스에서 예측한 a,b 와 실제 a,b의 차이를 계산하는건가요..? 그렇다면 colorization network는 정상인 데이터로 사전학습된건지 궁금합니다. 그리고 중간에 CAAN과 ADN이 지도학습인거 같다고 하셨는데 혹시 어느부분에서 그런걸까요..? Gt없이 학습하는 것 같아 질문드립니다
ΔCIEDE2000에 대해서는 https://onlinelibrary.wiley.com/doi/epdf/10.1002/col.20070 를 참고해주세요. 단순차이를 계산하는건 아니고 해당 논문에서도 위의 레퍼런스를 참조걸어서 설명합니다. colorization network는 정상데이터로 사전학습된게 맞습니다. CAAN, ADN 이 저도 처음엔 colorization network에서 나온 attention map을 pseudo-GT로 사용하는 비지도학습 기반이라고 생각하고 논문을 읽었는데, 그게아니라 attention map은 그냥 attention map으로만 사용하고 별도의 GT를활용해서 loss를 계산하는걸로 이해했습니다. 자기들도 해당부분이 약점이라고 생각해서인지 논문에서 설명을 미흡하게 한다고 느꼈는데요, 논문 초반에 supervision과 unsupervision을 혼재하여 파이프라인을 구성하였다고 언급하는걸로보아 CAAN/ADN이 supervision기반이라고 생각됩니다. if가 낮은 학회의 논문이라 그런지 많이 미흡하네요.